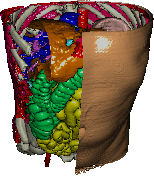
Medizinische Bildverarbeitung heute
Moderne bildgebende Verfahren wie z.B. die Computertomographie (CT) und Magnetresonanztomographie (MRT) ermöglichen, dreidimensionale Grauwertbilder der inneren Struktur von Objekten zu erstellen.
Die Tomographen erreichen dabei Auflösungen von 1mm³ oder höher und erzeugen somit sehr große Datenmengen. So belegen typische CT-Aufnahmen des menschlichen Abdomens typischerweise 400 Megabyte (Stapel von 800 Schichten mit je 512x512 Pixel Auflösung bei 2 Byte/Pixel zur Darstellung des großen Dynamikbereiches).
So große Datenmengen sind nicht mehr mit wirtschaftlichem Zeitaufwand durch schichtweise manuelle Beurteilung zu sichten und oft bleibt auch einem erfahrenen Betrachter mancher dreidimensionale Zusammenhang verborgen, der durch eine entsprechende Visualisierung sichtbar gemacht werden könnte.
Bis vor kurzem war es aufgrund der Speicher- und Rechenleistungsanforderungen jedoch nur auf aufwendiger Spezialhardware möglich, solche Visualisierungen durchzuführen. Und selbst wenn diese zur Verfügung stand, war die Verarbeitung in der Regel nicht echtzeitfähig und lieferte "offline" gerenderte Videosequenzen.
Das hier vorgestellte Programmpaket Eccet wurde entwickelt, um unter Einsatz kostengünstiger PC-Hardware, die inzwischen die notwendigen Voraussetzungen erfüllt, solche großen 3D-Datensätze aufzubereiten, in Echtzeit zu visualisieren und Hilfestellung bei der Interpretation zu geben.
Die Entwicklung eines eigenen Systems erschien sinnvoll, da bisher kommerziell verfügbare Lösungen weder preislich noch von der gewünschten Funktionalität, Flexibilität und Leistung in Frage kamen.
Eccet wurde zunächst als Visualisierungssystem entwickelt, um die Ergebnisse des am Institut für Informatik der Heinrich-Heine-Universität Düsseldorf entwickelten Filterverfahrens NLG plastisch darstellbar zu machen. Später wurden in Zusammenarbeit mit der Uniklinik Düsseldorf und anderen Forschungseinrichtungen Filter- und Segmentierfunktionen sowie Detektionsalgorithmen für medizinisch interessante Strukturen in das System integriert, so daß sich insgesamt ein in sich abgeschlossenes System zur Vorverarbeitung, Segmentierung und Visualisierung dreidimensionaler Datensätze ergibt.
Ein modularer Aufbau ermöglicht dabei, leicht Erweiterungen in das System einzubauen, sowie einzelne Teile auf mehrere Rechner zu verteilen, um mehr Information gleichzeitig darstellen zu können.
Eccet nutzt - soweit die eingesetzten Algorithmen dies erlauben - konsequent Multithreading um auf Multiprozessor-Architekturen die volle Leistungsfähigkeit ausnutzen zu können.
Virtuelle Koloskopie an der Universität Düsseldorf:
Eine Einführung in virtuelle Koloskopie der Klinik für Gastroenterologie, Hepatologie und Infektiologie
Mathematische Methoden in der virtuellen Koloskopie
Virtuelle CT-Kolographie mit der Mehrzeilen-Spiral-Computertomographie in Niedrig-Dosis-Technik: erste Ergebnisse